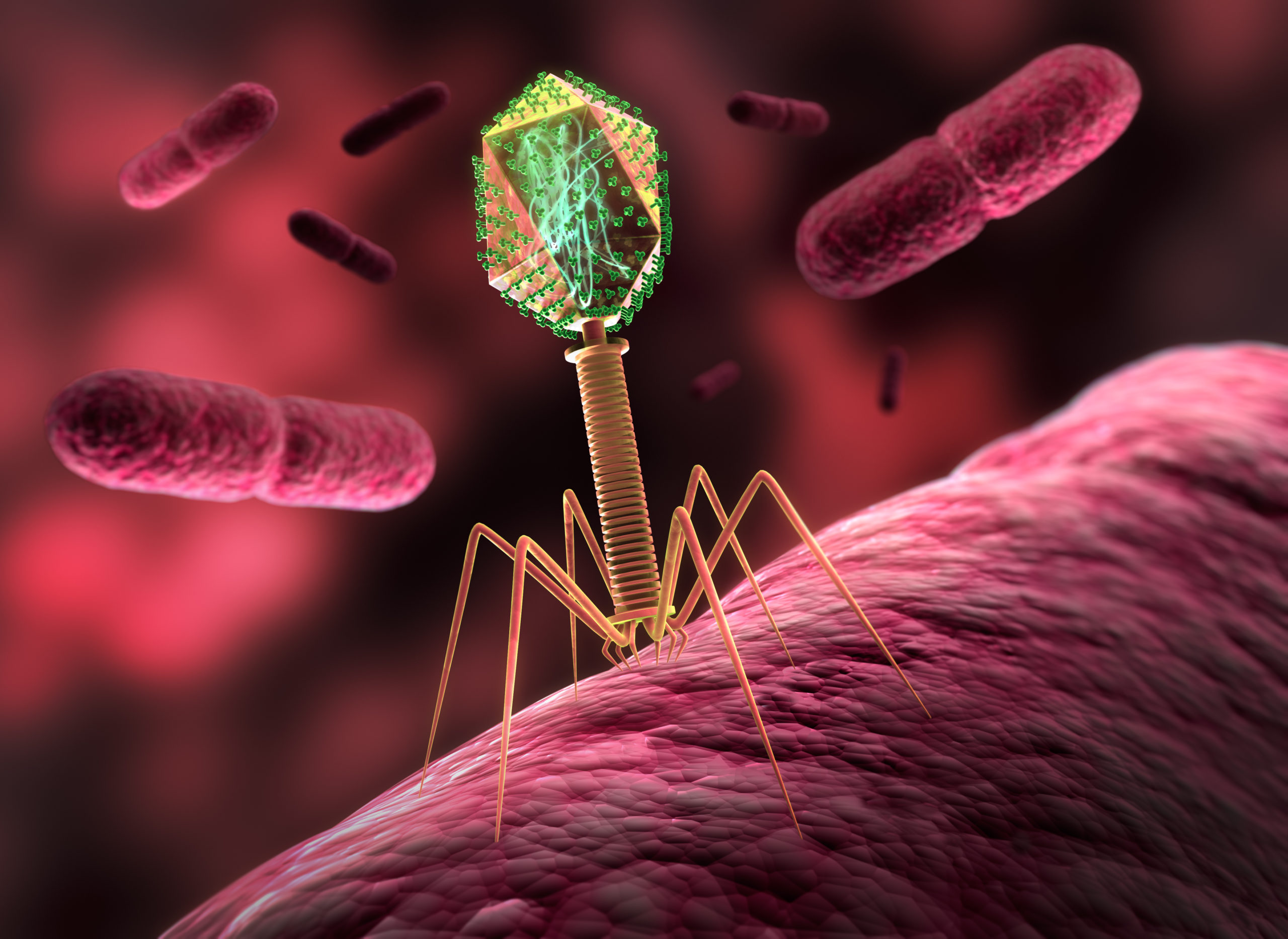
Tout comme les virus ont besoin de nos cellules pour se répliquer, les bactériophages ont besoin des bactéries qu’ils infectent pour se répliquer. Différents bactériophages ciblent ainsi différentes bactéries. Typiquement, les bactériophages se fixent à des récepteurs situés à la surface de certaines bactéries, et les infectent en y injectant leur matériel génétique. Les bactériophages dits “virulents” ont un cycle lytique, au cours duquel ils utilisent les ressources énergétiques de la bactérie infectée pour répliquer leur matériel génétique et former de nouveaux bactériophages (jusqu’à 1000 nouveaux bactériophages par bactérie) qui sont relargués en détruisant la bactérie. Les bactériophages dits “tempérés” ont un cycle lytique mais aussi un cycle lysogénique, durant lequel ils se répliquent en même temps que les bactéries infectées, sans les tuer.
La capacité des bactériophages à détruire certaines bactéries de façon sélective est connue depuis longtemps. Au cours du XXe siècle, des chercheurs ont ainsi utilisé des bactériophages comme des médicaments pour traiter des infections bactériennes comme la dysenterie. Toutefois, cette approche était à l’époque considérée comme étant moins fiable que les antibiotiques. La plupart des pays, à l’exception de ceux du bloc soviétique, ont donc abandonné la thérapie par bactériophages et privilégié l’utilisation d’antibiotiques. Aujourd’hui, bien que la thérapie par bactériophages ne soit pas autorisée en Europe ou aux USA, des bactériophages sont utilisés par l’industrie agroalimentaire pour contrôler l’absence de certaines bactéries dans les produits destinés à l’alimentation. Toutefois, le problème de résistance bactérienne aux antibiotiques a relancé l’intérêt pour une approche thérapeutique impliquant l’utilisation de bactériophages2.
Concernant le microbiome intestinal, les bactéries ont été davantage étudiées que les virus qui les infectent parce que c’est beaucoup plus facile. En effet, la caractérisation des bactéries repose en grande partie sur le fait que certains gènes bactériens sont très proches entre bactéries de même espèce mais distincts entre bactéries d’espèces différentes 3-4. Or, par rapport aux bactéries, les gènes des bactériophages ont une variabilité beaucoup plus élevée 5. Autrement dit, deux bactériophages “identiques” sont bien souvent génétiquement beaucoup plus différents que deux bactéries “identiques”. Par exemple, les séquences génomiques de tous les bactériophages recensés (quelques milliers) ne présentent aucune homologie entre elles dans 97% des cas 6. Et s’il est possible de mettre un nom sur les bactéries présentes dans un échantillon donné à partir de tout l’ADN présent dans cet échantillon, ce n’est pas possible avec les bactériophages. Ainsi, 75 à 99 % des séquences d’ADN de bactériophages ne correspondent à aucune séquence d’ADN viral connu7.
Malgré ces difficultés, des méthodes ont été développées pour caractériser les bactériophages de l’intestin et un premier virome humain (ou “phageome”) a ainsi été publié en 2003 8. On estime qu’il y a près de 108 à 109 bactériophages par gramme de feces soit à peu près 1012 bactériophages dans l’intestin humain 5,9. Comme pour les bactéries, la composition intestinale en bactériophages fluctue durant les deux premières années puis reste relativement stable au cours du temps tout en étant influencée par le régime alimentaire 10-14. La majorité de ce virome intestinal est identique entre individus sains, composé essentiellement de bactériophages virulents comme Caudovirales et Microviridae, et seule une minorité de bactériophages est propre à chacun 15,16.
Alors après tout, si les bactéries se font infecter par des bactériophages dans notre intestin, en quoi cela nous concerne-t-il ? Et bien tout simplement parce que les bactériophages agissent comme des régulateurs des populations bactériennes qu’ils infectent. En passant de bactéries en bactéries, les bactériophages apportent de nouveaux gènes et contribuent à la diversification des populations bactériennes 17. Et les bactériophages peuvent avoir des effets néfastes ou bénéfiques pour notre santé. Un exemple d’effet néfaste est que des bactériophages peuvent transporter des gènes codant des toxines de bactéries pathogènes à des bactéries non pathogènes. Un exemple d’effet bénéfique est que la colonisation de la couche de mucus surplombant l’épithélium intestinal par des bactériophages constitue une protection, qui s’ajoute à la protection immunitaire (anticorps et peptides antibactériens) contre des bactéries qui auraient des velléités invasives 18-19.
Et si les virus contribuent à l’équilibre bactérien qui est si important pour rester en bonne santé, détecte-t-on des déséquilibres viraux dans les maladies liées au microbiome intestinal ? La réponse est oui. Chez les patients atteints de maladies inflammatoires chroniques de l’intestin (comprenant la maladie de Crohn et la rectocolite hémorragique), la composition en bactériophages est différente 15,20. Alors que ces patients on une diversité bactérienne réduite, ils ont un enrichissement de la diversité en bactériophages et plus particulièrement en Caudovirales, et également une augmentation du ratio bactériophages tempérés / bactériophages virulents par rapport à des individus sains 21,22.
Le rôle probable des bactériophages dans la régulation des populations bactériennes de l’intestin et le fait que la composition en bactériophages soit associée à des pathologies intestinales soulève le rôle possible des bactériophages de façon plus large. L’efficacité stupéfiante du transfert de matière fécale (FMT, pour fecal microbial transplant) pour traiter les infections intestinales par la bactérie Clostridium difficile a nourri l’espoir qu’une telle approche puisse potentiellement être efficace pour d’autres pathologies intestinales 23–26. Or on considère généralement que la FMT fonctionne en remplaçant un microbiome bactérien défectueux par un microbiome bactérien sain 27. Seulement, le transfert de matière fécale n’est généralement pas un transfert de bactéries intestinales mais un transfert de tout le reste également. Ainsi, une étude a montré que la FMT fonctionne tout aussi bien si l’on retire les bactéries des feces transférées au receveur, soulevant la question du rôle des bactériophages intestinaux dans l’efficacité de cette approche 28. Les bactériophages pourraient également jouer un rôle dans la réponse aux traitements anticancéreux. Le cyclophosphamide, une molécule largement utilisée en chimiothérapie, induit le passage de la bactérie intestinale E.hirae de l’intestin vers des organes lymphoïdes où elle induit une réponse immunitaire pouvant cibler des cellules tumorales. Le groupe de Laurence Zitvogel a démontré que ce n’étaient pas ces bactéries qui activaient une réponse immunitaire anticancéreuse spécifique, mais un bactériophage situé à l’intérieur de ces bactéries dont des molécules étaient très proches de celles exprimées par des cellules cancéreuses 29.
Le microbiome intestinal est donc bien plus que la simple somme des bactéries de l’intestin et le développement de nouveaux outils et de nouvelles méthodes va probablement permettre de mieux comprendre l’impact que peuvent avoir les bactériophages de l’intestin sur la santé dans les prochaines années.
Références
- W. B. Whitman, D. C. Coleman et W. J. Wiebe. “Prokaryotes : the unseen majority”. In : Proc Natl Acad Sci U S A 95.12 (juin 1998), p. 6578-6583.
- D. M. Lin, B. Koskella et H. C. Lin. “Phage therapy : An alternative to antibiotics in the age of multi-drug resistance”. In : World J Gastrointest Pharmacol Ther 8.3 (août 2017), p. 162-173.
- M. H. Fraher, P. W. O’Toole et E. M. Quigley. “Techniques used to characterize the gut microbiota : a guide for the clinician”. In : Nat Rev Gastroenterol Hepatol 9.6 (mar. 2012), p. 312-322.
- D. J. Lane et al. “Rapid determination of 16S ribosomal RNA sequences for phylogenetic analyses”. In : Proc Natl Acad Sci U S A 82.20 (oct. 1985), p. 6955-6959.
- M. B. Dion, F. Oechslin et S. Moineau. “Phage diversity, genomics and phylogeny”. In : Nat Rev Microbiol 18.3 (mar. 2020), p. 125-138.
- T. N. Mavrich et G. F. Hatfull. “Bacteriophage evolution differs by host, lifestyle and genome”. In : Nat Microbiol 2 (juil. 2017), p. 17112.
- V. Aggarwala, G. Liang et F. D. Bushman. “Viral communities of the human gut : meta- genomic analysis of composition and dynamics”. In : Mob DNA 8 (2017), p. 12.
- M. Breitbart et al. “Metagenomic analyses of an uncultured viral community from human feces”. In : J Bacteriol 185.20 (oct. 2003), p. 6220-6223.
- A. N. Shkoporov et C. Hill. “Bacteriophages of the Human Gut : The « Known Unknown » of the Microbiome”. In : Cell Host Microbe 25.2 (fév. 2019), p. 195-209.
- E. S. Lim et al. “Early life dynamics of the human gut virome and bacterial microbiome in infants”. In : Nat Med 21.10 (oct. 2015), p. 1228-1234.
- A. Reyes et al. “Viruses in the faecal microbiota of monozygotic twins and their mothers”. In : Nature 466.7304 (juil. 2010), p. 334-338.
- S. Minot et al. “Rapid evolution of the human gut virome”. In : Proc Natl Acad Sci U S A 110.30 (juil. 2013), p. 12450-12455.
- A. N. Shkoporov et al. “The Human Gut Virome Is Highly Diverse, Stable, and Individual Specific”. In : Cell Host Microbe 26.4 (oct. 2019), p. 527-541.
- S. Minot et al. “The human gut virome : inter-individual variation and dynamic response to diet”. In : Genome Res 21.10 (oct. 2011), p. 1616-1625.
- P. Manrique et al. “Healthy human gut phageome”. In : Proc Natl Acad Sci U S A 113.37 (sept. 2016), p. 10400-10405.
- A. N. Shkoporov et al. “Reproducible protocols for metagenomic analysis of human faecal phageomes”. In : Microbiome 6.1 (avr. 2018), p. 68.
- R. Sausset et al. “New insights into intestinal phages”. In : Mucosal Immunol 13.2 (mar. 2020), p. 205-215.
- G. M. F. Almeida et al. “Bacteriophage Adherence to Mucus Mediates Preventive Protection against Pathogenic Bacteria”. In : mBio 10.6 (nov. 2019).
- J. J. Barr et al. “Bacteriophage adhering to mucus provide a non-host-derived immunity”. In : Proc Natl Acad Sci U S A 110.26 (juin 2013), p. 10771-10776.
- T. Zuo et al. “Gut mucosal virome alterations in ulcerative colitis”. In : Gut 68.7 (juil. 2019), p. 1169-1179.
- J. M. Norman et al. “Disease-specific alterations in the enteric virome in inflammatory bowel disease”. In : Cell 160.3 (jan. 2015), p. 447-460.
- A. G. Clooney et al. “Whole-Virome Analysis Sheds Light on Viral Dark Matter in Inflam- matory Bowel Disease”. In : Cell Host Microbe 26.6 (déc. 2019), p. 764-778.
- J. S. Bakken. “Fecal bacteriotherapy for recurrent Clostridium difficile infection”. In : Anae- robe 15.6 (déc. 2009), p. 285-289.
- E. van Nood et al. “Duodenal infusion of donor feces for recurrent Clostridium difficile”. In : N Engl J Med 368.5 (jan. 2013), p. 407-415.
- T. J. Borody et A. Khoruts. “Fecal microbiota transplantation and emerging applications”. In : Nat Rev Gastroenterol Hepatol 9.2 (déc. 2011), p. 88-96.
- L. P. Smits et al. “Therapeutic potential of fecal microbiota transplantation”. In : Gastroen- terology 145.5 (nov. 2013), p. 946-953.
- A. Khoruts et M. J. Sadowsky. “Understanding the mechanisms of faecal microbiota trans- plantation”. In : Nat Rev Gastroenterol Hepatol 13.9 (sept. 2016), p. 508-516.
- T. Zuo et al. “Bacteriophage transfer during faecal microbiota transplantation in Clostridium difficile infection is associated with treatment outcome”. In : Gut 67.4 (avr. 2018), p. 634-643.
- A. Fluckiger et al. “Cross-reactivity between tumor MHC class I-restricted antigens and an enterococcal bacteriophage”. In : Science 369.6506 (août 2020), p. 936-942.
Auteur

Julien Verdier


Merci pour cet article très enrichissant. Ne pas oublier également les champignons qui interagissent avec tout ce petit monde!